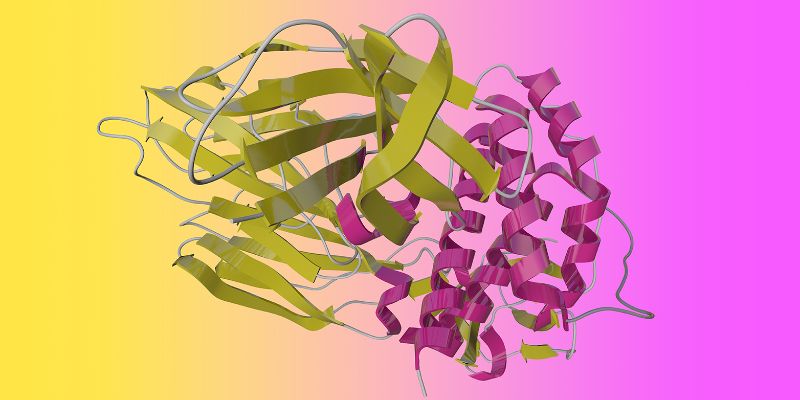
Les algorithmes informatiques révolutionnent la biologie
Les algorithmes informatiques sont un outil utile pour la recherche biomédicale depuis des décennies, et leur importance n'a cessé de croître au fil du temps. Mais ce que nous vivons aujourd'hui n'est rien de moins qu'un saut quantique; il éclipse tout ce qui a précédé et aura des effets imprévus. Les algorithmes d'intelligence artificielle (IA) ont permis d'utiliser uniquement la séquence linéaire des éléments constitutifs des protéines - les acides aminés - pour fournir des prédictions extrêmement précises sur la structure tridimensionnelle dans laquelle cette chaîne d'acides aminés s'assemblera.
Pour saisir l'importance de ce développement, il faut savoir que la biologie au niveau cellulaire est en fait toujours une question d'interactions spatiales entre molécules, et que c'est la structure tridimensionnelle de ces molécules qui détermine ces interactions. Une fois que nous comprenons les structures et les interactions en jeu, nous comprenons la biologie. Et ce n'est que lorsque nous comprenons la structure des molécules que nous pouvons concevoir des médicaments capables d'influencer la fonction de ces molécules.
Jusqu'à présent, il existait trois méthodes expérimentales pour déterminer la structure tridimensionnelle des protéines: l'analyse de la structure par rayons X, la résonance magnétique nucléaire et, depuis quelques années seulement, la microscopie cryo-électronique. L'ajout maintenant de l'IA comme quatrième méthode de précision n'est pas seulement dû aux améliorations des algorithmes de l'IA et à la vaste puissance de calcul disponible aujourd'hui. Pour que l'IA puisse faire des prédictions précises, elle doit également être entraînée à l'aide d'une multitude de données d'une qualité exceptionnelle. Ce qui rend possible le saut quantique susmentionné, ce sont des progrès et des efforts considérables, tant dans la science des données que dans la recherche expérimentale sur les protéines.
La concurrence entre la recherche privée et la recherche publique
Le programme d'IA AlphaFold, développé par DeepMind, une société sœur de Google, occupe actuellement le devant de la scène. À l'heure actuelle, DeepMind est sans aucun doute l'acteur le plus important dans la prédiction des structures protéiques. Mais ce qui se perd dans le débat public, c'est que DeepMind est loin d'être le seul acteur dans ce domaine; l'équipe dirigée par David Baker de l'Université de Washington, en particulier, mène des recherches remarquables.
Dans l'ensemble, cette concurrence entre la recherche privée et la recherche publique a sûrement servi à inspirer et à revigorer le domaine, même si, comme on peut s'y attendre, les acteurs privés gardent pour eux bon nombre de leurs idées afin de protéger leurs propres intérêts commerciaux. Mais la recherche hautement compétitive a également permis d'améliorer considérablement les algorithmes d'IA qui sont dans le domaine public et que l'ensemble de la communauté scientifique peut désormais utiliser et développer. Je pense que cette tendance va se poursuivre. Les algorithmes d'IA nous fourniront bientôt des structures très précises pour toutes les protéines connues. Cela nous permettra de concevoir des médicaments de précision sur ordinateur.
À l'avenir, il devrait être possible de partir d'un échafaudage moléculaire tridimensionnel conçu sur un ordinateur et d'utiliser l'IA pour calculer une séquence d'acides aminés qui s'assembleront précisément dans la structure souhaitée avec la fonction moléculaire désirée.
Une fois que cette séquence d'acides aminés a été déterminée, mon domaine de recherche entre en jeu. Mon travail porte sur le développement de gènes et de génomes artificiels, et il fait également appel à des algorithmes informatiques. Sur la base des séquences d'acides aminés, nous calculons comment l'information sur les protéines peut être codée dans des séquences de blocs de construction génétiques - en d'autres termes, dans l'ADN. Et nous le faisons d'une manière qui fournit un moyen simple de synthétiser ces gènes pour des applications pratiques.
Inverser le flux d'informations
Cela signifie que nous sommes sur le point de pouvoir calculer un gène artificiel pour toute structure protéique tridimensionnelle donnée conçue sur ordinateur, puis de synthétiser ce gène. En biotechnologie, cela ouvre la voie à la fabrication de protéines artificielles dans des micro-organismes - y compris de nouveaux agents pharmaceutiques, vaccins ou enzymes destinés à l'industrie.
Depuis l'apparition des premières formes de vie il y a plusieurs milliards d'années, les informations biologiques ont toujours été stockées sous forme d'ADN. À l'intérieur des cellules biologiques, ces informations sont transcrites, d'abord en molécules d'ARN, puis traduites en protéines. Jusqu'à présent, il n'existait aucun mécanisme permettant d'inverser le flux d'informations, de sorte que les informations protéiques soient retraduites en informations ADN. L'IA va bientôt changer tout cela. Pour les biologistes comme moi, il s'agit d'un développement incroyablement spectaculaire, qui aura un impact profond sur la biotechnologie et la médecine.