Tiere mithilfe von Deep Learning verfolgen
Das Verhalten von Tieren zu erfassen, ist für die Neurowissenschaft und Ökologie sowie viele weitere Fachbereiche sehr wichtig. Obwohl Kameras ohne Probleme differenzierte Bewegungen von Tieren erfassen können, ist es schwierig, einem Computer beizubringen das Verhalten von Tieren aus den visuellen Informationen herauslesen können. Es ist interessant, dass unser eigenes «visuelles System» das scheinbar mühelos kann.
Eine der wichtigsten Komponenten bei der Quantifizierung von Tierverhalten ist die «Körperteilerkennung» (pose estimation). Ein Computer muss dazu die Position und Ausrichtung verschiedener Körperteile eines Tiers identifizieren können. In einer Laborumgebung können Forschende Reflektoren am Körper des Tiers anbringen, um präzisere Ergebnisse zu erhalten. Diese Bewegungserfassungstechnik kommt auch auf Filmsets (wie bei Gollum von Der Herr der Ringe) zum Zug. Wie man sich aber vorstellen kann, ist es nicht einfach, Tiere mit spezieller Ausrüstung auszustatten und solche Reflektoren beeinträchtigen das Verhalten. In der freien Natur ist diese Praktik geradezu unmöglich und würde zudem gegen ethische Prinzipien verstossen.
Aus diesem Grund haben die Professoren Alexander Mathis und Mackenzie Mathis Methoden zum «markierungslosen» Tracking von Tieren erforscht. Die Software des Forschungsteams stützt sich auf Deep Learning, um Computern beizubringen, wie sie alle Körperteile ohne physische oder visuelle Markierungen schätzen können.
Ihre Teams haben DeepLabCut entwickelt, ein Open-Source-Paket zur Körperteilerkennung von Tieren. Dank Deep Learning kann die Software die Bewegungen von Tieren ohne Markierungen erfassen. Die Software hat seit der Einführung im Jahr 2018 im Fachbereich der Biowissenschaften signifikante Popularität erlangt: über 350 000 Softwaredownloads und fast 1400 Literaturerwähnungen. 2020 entwickelten die Teams um das Duo Mathis DeepLabCut-Live!. Es ist eine Real-Time-Version von DeepLabCut mit minimaler Latenzzeit. Forschende können mit der Live-Version Tierbewegungen in Echtzeit analysieren und den Tieren verhaltensspezifisches Feedback geben.
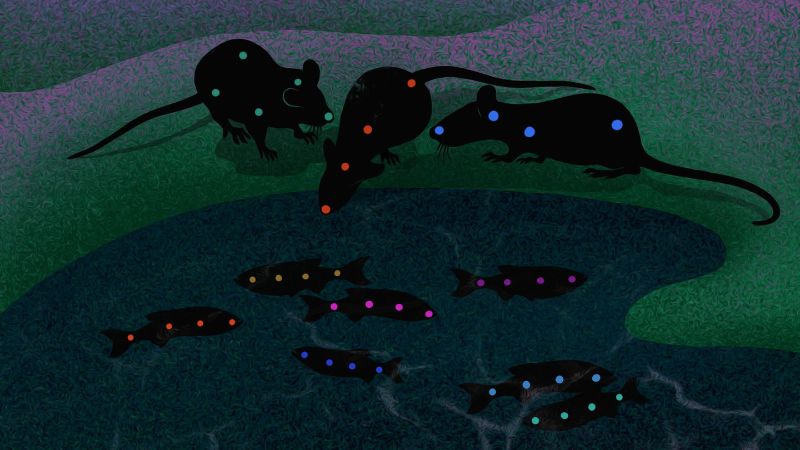
Zwei Jahre später ist nun eine Erweiterung von DeepLabCut erschienen, die das Bewegungstracking von Gruppen von Tieren verbessert. Bislang war es schwierig, die Posen von Tieren in Gruppen zu schätzen, da sie sich oft nahe beieinander bewegen, wie z.B. in Fischschwärmen oder wenn sich Mäuse um ihre Jungtiere kümmern. Die Herausforderungen liegen auf der Hand: Die einzelnen Tiere sehen einander so ähnlich, dass sie der Computer verwechselt, und die Tiere können sich gegenseitig verdecken. Zudem möchten Forschende unter Umständen viele computergenerierte Punkte oder «Keypoints» verfolgen können, was eine effiziente Datenverarbeitung erschwert.
Die Forschenden stellten sich der Herausforderung, indem sie zuerst vier unterschiedlich komplexe Datensätze erstellten, um neue Methoden zu testen. Die Datensätze, die zusammen mit Kolleginnen und Kollegen am MIT und der Harvard Universität erhoben wurden, umfassen drei Mäuse; eine Maus mit Nachwuchs; Paare von Seidenäffchen in einem grossen Gehege und 14 Fische in einer Strömungswanne. Anhand dieser Datensätze konnten die Forschenden neue Methoden entwickeln, um den zuvor genannten Schwierigkeiten zu begegnen.
DeepLabCut wurde daraufhin in drei Bereichen erweitert: neuartige Netzwerkarchitekturen, datengesteuerte Assoziationsverfahren (welche erkannte Körperteile zu den jeweiligen Individuen verknüpfen) und anpassbare Methoden zum tracken. Konkret haben die Forschenden ein neues neuronales Multi-Task-Netzwerk entwickelt, das aus einzelnen Bildern Körperteile, Gliedmassen und die Identität des Tieres ableiten kann. Ausserdem entwickelten sie einen vom Körperbau der Tiere unabhängiges Assoziationsverfahren. Das ist wichtig, wenn man mit Tieren arbeitet, die sich in ihrer Körperform stark unterscheiden können. Diese Methoden wurden an Körperbauplänen von Fischen bis hin zu Primaten validiert.
Darüber hinaus entwickelten die Forschenden eine Methode zur Identifizierung einzelner Tiere anhand von Videoaufnahmen ohne Ground Truth Daten. «Man stelle sich vor, wie schwierig es ist, zuverlässig festzustellen, welche Labormaus welche ist», meint Mackenzie Mathis. «Sie sehen einander so ähnlich, dass eine Unterscheidung mit blossem Auge fast unmöglich ist.»
Der neue Algorithmus basiert auf metrischem Lernen mit Transformatoren zur Bilderkennung. Wenn zum Beispiel mehrere Tiere aus dem Blickfeld verschwinden und später wieder auftauchen, kann der Algorithmus bereits identifizierte Tiere erneut erkennen und weiterverfolgen. Ein ähnlicher Ansatz, der auf dem Aussehen der Tiere beruht, kam bei der Verhaltensstudie von Seidenäffchenpaaren zur Anwendung. Die Analyse von 9h Verhaltensdaten ergab unter anderem, dass Seidenäffchen, eine sehr soziale Spezies, gerne gemeinsam in ähnliche Richtungen schauen.
«DeepLabCut wird in hunderten von Laboren auf der ganzen Welt verwenden. Wissenschaftlerinnen und Wissenschaftler haben damit alles mögliche analysiert, von der Mimik bei Mäusen bis zum Greifverhalten von Primaten», sagt Alexander Mathis. «Ich bin wirklich gespannt, zu sehen, was die Community mit den erweiterten Funktionen zur Analyse sozialer Interaktionen anstellen wird.»