Satelliten-DNA spielt Schlüsselrolle bei Artbildung
Mehr als 10 Prozent unseres Genoms bestehen aus sich wiederholenden, scheinbar unsinnigen Abschnitten genetischen Materials, das als Satelliten-DNA bezeichnet wird. Diese trägt nicht einmal Baupläne für Proteine. Einige Wissenschaftler haben sie deshalb als «genetischen Müll» bezeichnet.
In einer Reihe von Arbeiten haben Madhav Jagannathan, zurzeit Assistenzprofessor am Departement Biologie der ETH Zürich, und seine frühere Postdoc-Betreuerin Yukiko Yamashita vom Whitehead Institute am MIT (USA) jedoch nachgewiesen, dass Satelliten-DNA kein Müll ist, sondern in Zellen eine wesentliche Rolle spielt: Sie arbeitet mit Proteinen in der Zelle zusammen, um alle Chromosomen einer Zelle in ihrem Kern zusammenzuhalten.
In ihrer jüngsten Arbeit, die vor kurzem in der Fachzeitschrift «Molecular Biology and Evolution» veröffentlicht wurde, gehen Jagannathan und Yamashita noch einen Schritt weiter. Sie schlagen vor, dass das System der Chromosomenorganisation durch die Satelliten-DNA ein wichtiger Grund dafür ist, dass Organismen verschiedener Arten keine lebensfähigen Nachkommen hervorbringen können.
Der Ursprung der Arten: DNA-Ausgabe
Dass die Satelliten-DNA zwischen den Arten sehr variabel ist, ist seit Jahren bekannt. «Wenn man das Schimpansen-Genom mit dem menschlichen Genom vergleicht, sind die proteinkodierenden Regionen zu 98 bis 99 Prozent identisch», sagt Yamashita. «Aber die Junk-DNA der beiden Arten unterscheidet sich stark.»
Dabei handelt es sich bei der Satelliten-DNA um die Sequenzen im Genom, die sich am schnellsten verändern. «Die bisherige Sichtweise ist, dass es ‘nur’ genetischer Müll ist, bei dem es keine Rolle spielt, ob er sich von Art zu Art unterscheidet», erklärt der Assistenzprofessor am Departement Biologie der ETH.
Als er und seine Kollegin jedoch die Bedeutung der Satelliten-DNA für die Fruchtbarkeit und das Überleben «reiner» Arten untersuchten, erhielten sie den ersten Hinweis darauf, dass diese Sequenzen eine Rolle bei der Artbildung spielen könnten.
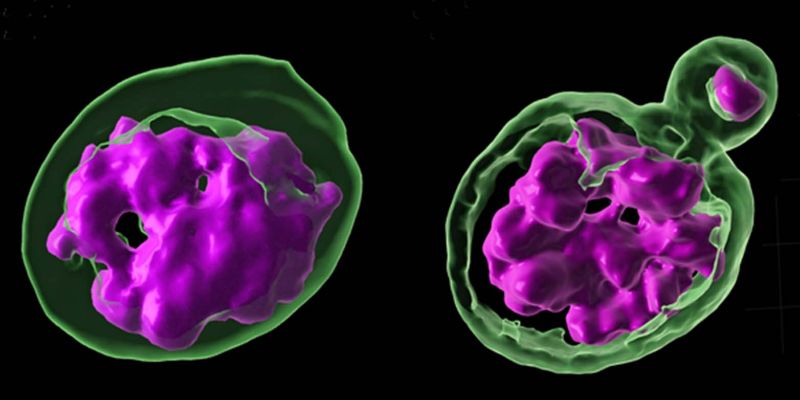
In einem ihrer Versuche schalteten die Forschenden in einer Taufliege Drosophila melanogaster ein Protein namens «Prod» aus. In der Folge zerstreuten sich die Chromosomen der Fliegen ausserhalb des Zellkerns und lagerten sich in winzigen Klumpen zellulären Materials ab. Die Fliegen starben. Normalerweise bindet «Prod» an eine bestimmte Stelle der Satelliten-DNA dieser Taufliege. «Dieses Stück Satelliten-DNA, an welches sich das Prod-Protein andockt, fehlt in den nächsten Verwandten von Drosophila melanogaster komplett», sagt Jagannathan.
Wenn also dieses spezifische Stück Satelliten-DNA bei der einen Art überlebenswichtig ist, bei der anderen aber fehlt, könnte dies folgendes bedeuten: dass diese zwei Fliegenarten im Laufe der Zeit unterschiedliche Satelliten-DNA-Sequenzen für dieselbe Aufgabe – die Chromosomenorganisation - entwickelt hatten. Jagannathan und Yamashita fragten sich deshalb, ob diese evolutiv bedingten Unterschiede ein Grund dafür sein könnten, dass verschiedene Arten sich nicht miteinander fortpflanzen können.
Eine Geschichte von zwei Fruchtfliegenarten
Um zu untersuchen, wie die Unterschiede in der Satelliten-DNA die reproduktive Inkompatibilität beeinflussen, konzentrierten sich die Forschende auf Drosophila melanogaster und deren nächste Verwandte, Drosophila simulans. Diese beiden Arten haben sich vor rund zwei Millionen Jahren aufgetrennt.
Wissenschaftler können zwar ein Drosophila melanogaster-Weibchen mit einem Drosophila simulans-Männchen kreuzen, aber die Nachkommen sind entweder steril oder sie sterben.
Jagannathan und Yamashita untersuchten dann das Gewebe dieser Hybriden, um herauszufinden, was sie sterben liess. Dabei fiel den Forschenden rasch etwas Interessantes auf: Das Gewebe von Hybriden zeigte die exakt gleichen Merkmale wie Gewebe von Fliegen «reiner» Arten mit gestörter Satelliten-DNA: die Chromsomen lagen bei beiden Formen verstreut in Zellen vor und nicht im Kern gebündelt.
Die Forschenden konnten jedoch eine gesunde Hybridfliege erzeugen, indem sie bestimmte Gene, die sogenannten Hybrid-Inkompatibilitätsgene, in den Elternfliegen veränderten. Diese Gene lassen sich an der Satelliten-DNA in den Zellen reiner Arten nachweisen und beeinflussten im Experiment die Chromosomenverpackung in Hybriden.
Insgesamt deuten diese Ergebnisse darauf hin, dass sich die Proteine, welche die Satelliten-DNA binden, ebenso rasch wandeln müssen wie die Satelliten-DNA, die sich rasant verändert. Nur so kann das Bindeprotein die korrekte Organisation der Chromosomen im Zellkern gewährleisten. Jede Art entwickelt im Lauf der Zeit ihre eigene «Strategie» für den Umgang mit der Satelliten-DNA. Kreuzen sich zwei verwandte Arten mit unterschiedlichen Strategien, werden die Chromosomen ausserhalb des Zellkerns verstreut.
In zukünftigen Studien wollen Jagannathan und Yamashita ihr Modell weiter testen: Wenn es ihnen gelingt, ein Protein zu entwerfen, das die Satelliten-DNA zweier verschiedener Arten binden kann, könnten sie theoretisch eine lebensfähige Kreuzung erhalten, die auch Nachkommen hervorbringen kann. Um dies zu bewerkstelligen, sind aber noch Jahre weiterer Forschung nötig.
Dieser Text von Eva Frederick vom Whitehead Institute, MIT, erschien zuerst hier.